Em mais um tópico da coluna Drops de Genômica, o oncologista André Murad (foto) explica a análise de fragmentos na detecção da mutação de sítio "splicing" do exon 14 do gene MET. Confira.
Em mais um tópico da coluna Drops de Genômica, o oncologista André Murad (foto) explica a análise de fragmentos na detecção da mutação de sítio "splicing" do exon 14 do gene MET. Confira.
Por André Murad*
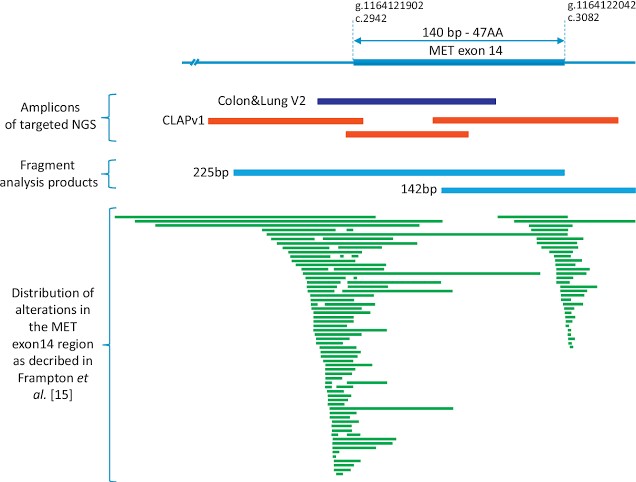
A comparação de duas técnicas usadas na triagem de amostras de tumor de câncer de pulmão de células não pequenas (CPCNP) demonstrou que a análise de fragmentos pode detectar grandes deleções do sítio de emenda ("splicing") do exon 14 de MET não identificadas pelo sequenciamento de próxima geração (NGS). Os resultados foram apresentados no Congresso de Análise Molecular para a Oncologia de Precisão (MAP) de 2020 (Abstract4MO).
Uma equipe do Departamento de Oncologia Molecular do Hôpital Européen Georges-Pompidou – APHP, em Paris, conduziu este estudo para avaliar a utilidade do NGS em comparação com a análise de fragmentos através da técnica de Sequenciamento Sanger e Análise de Fragmentos por Eletroforese Capilar para a detecção de alterações no exon 14 de MET, incluindo as mutações em sítio "splicing" que atuam como um alvo oncogênico e são observadas em 1% a 4% dos CPCNP.
O capmatinibe foi recentemente aprovado pela Food and Drug Administration para pacientes adultos com CPCNP metastático e mutações "splicing" do exon 14 de MET, tornando necessária a detecção de alterações de MET para identificar os candidatos ao tratamento. Além disso, as mutações do exon 14 de MET podem servir como um marcador de baixas taxas de resposta ao tratamento com terapia com inibidor de checkpoint imunológico (anti-PD1, anti-PDL1), apesar da alta expressão de PD-L1 tumoral.
Uma proporção substancial de CPCNP's que abrigam alterações do Exon 14 do gene MET são expressores de PD-L1. Apesar da expressão de PD-L1 frequente, as respostas à imunoterapia foram em geral incomuns e inferiores às observadas com terapia direcionada para este subconjunto genomicamente definido de pacientes com câncer de pulmão.
Comparação da eficácia do método
Os investigadores examinaram 87 amostras de CPCNP avançado. As amostras foram obtidas em exames de rotina dentro de um período de 18 meses e eram negativas para outros alvos moleculares testados. A mutação "splicing" do exon 14 de MET foi rastreada por análise de fragmento para detectar grandes deleções no exon 14 de MET, e por NGS em uma plataforma de Ion Proton usando a tecnologia Ampliseq. Grandes deleções detectadas apenas por análise de fragmento também foram caracterizadas por sequenciamento de Sanger. A expressão do tumor PD-L1 foi quantificada por imunohistoquímica.
Das 87 amostras de pacientes avaliadas por análise de fragmento e sequenciamento, uma mutação "splicing" do exon 14 do MET foi detectada em 13 pacientes. Entre os 13 pacientes, oito amostras tiveram deleção impactando o local de emenda e cinco tiveram variantes de emenda. Entre as oito deleções, duas eram grandes deleções de 41 bp e 66 bp. Essas duas deleções foram determinadas apenas por análise de fragmento e não foram detectadas no NGS. No entanto, foi observada uma associação com a expressão de PD-L1; as amostras com mutação "splicing" do exon 14 de MET apresentaram valores mais elevados de PD-L1 do que outros tumores rastreados neste estudo.
O estudo concluiu que seus resultados apontam limitações da técnica de NGS para analisar grandes deleções do Exon -4 do gene MET e o consequente risco de resultados falsos negativos do teste. A associação de métodos complementares ou a triagem intrônica de ampla cobertura podem ser usadas alternativamente para que a detecção da mutação "splicing" do Exon 14 do gene MET tenha maior acurácia, o que contribui sobremaneira para a correta opção terapêutica com agentes anti-MET.