Em mais um tópico da coluna ‘Drops de Genômica’, o oncologista André Murad (foto) explora novos biomarcadores baseados em neoantígenos frameshift e sua relação com a avaliação de resposta e toxicidade aos inibidores de checkpoint no câncer de pulmão.
Em mais um tópico da coluna ‘Drops de Genômica’, o oncologista André Murad (foto) explora novos biomarcadores baseados em neoantígenos frameshift e sua relação com a avaliação de resposta e toxicidade aos inibidores de checkpoint no câncer de pulmão.
Por André Marcio Murad*
Estudos recentes têm avaliado uma fonte distinta de biomarcadores específicos do câncer: anticorpos para neoantígenos gerados pela tradução de erros no compartimento transcriptômico (do RNA). Em todas as células, os erros na transcrição e processamento do RNA ocorrem mais de 100 vezes mais frequentemente do que os erros na replicação do DNA. As taxas de erro em tumores são ainda maiores em relação às células saudáveis, e os sistemas de reparo normais são comprometidos ou sobrecarregados. Consequentemente, muitos desses erros de RNA são traduzidos em variantes de peptídeos com frameshift (ou com mudança na matriz de leitura). Por exemplo, o salto de exon durante o splicing pode criar transcrições que resultam em estruturas alternativas de codificação de proteínas. Indels resultantes do deslizamento da RNA polimerase através de loci de microssatélites também podem criar transcrições que levarão à tradução de estruturas alternativas de codificação de proteínas. A jusante dessas transcrições variantes, os aminoácidos incorretos criam caudas de proteína C-terminal aberrantes. Como estes são neoantígenos, não autoantígenos, eles são altamente propensos a provocar respostas de células T e B. Demonstrou-se que os neoantígenos frameshift (FSN) decorrentes do processamento incorreto do RNA em células tumorais geram peptídeos altamente imunogênicos.
Um estudo recente avaliou esse novo biomarcador para prever respostas tumorais e eventos imunológicos adversos a terapias com inibidores de checkpoint imunológico (ICI) em pacientes com câncer de pulmão avançado.
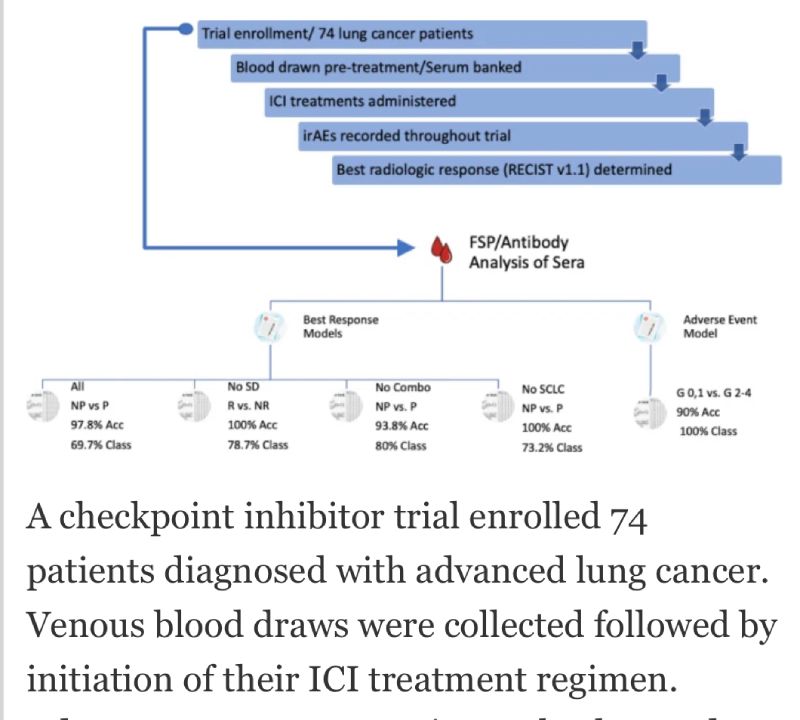
Amostras de soro foram obtidas de 74 pacientes com câncer de pulmão antes de terapias paliativas de PD-(L)1 com respostas tumorais subsequentemente registradas e eventos adversos imunes (irAEs). Amostras de pré-tratamento foram analisadas em microarranjos de peptídeos frameshift (FSPs), representando ~ 375.000 peptídeos variantes que as células tumorais podem ser previstas informaticamente para produzir a partir de erros de processamento de mRNA traduzidos.
As amostras de soro de câncer de pulmão foram classificadas com base em modelos preditivos dos resultados do tratamento com ICI. A progressão da doença foi prevista antes do tratamento com precisão de ~ 98% na coorte completa de todas as categorias de resposta, embora ~ 30% das amostras fossem indeterminadas.
Este modelo foi construído com uma coorte de amostra heterogênea de pacientes que (i) mostrariam uma resposta clara ou resultados estáveis, (ii) seriam administradas terapias únicas ou combinadas e (iii) foram diagnosticados com diferentes subtipos de câncer de pulmão. A remoção da doença estável, terapia combinada ou grupos de câncer de pequenas células da construção do modelo aumentou a proporção de amostras classificadas enquanto o desempenho permaneceu alto.
Análises informáticas mostraram que vários dos FSPs no modelo de todas as respostas foram mapeados para traduções de mRNAs variantes dos mesmos genes. No modelo preditivo para toxicidades de tratamento, a ligação aos FSPs associados ao irAE forneceu 90% de precisão no pré-tratamento, sem indeterminados. Vários dos FSPs de classificação exibiram similaridade de sequência com autoproteínas.
Em conclusão, anti-FSP podem servir como biomarcadores para prever resultados de ICI quando testados contra ligantes correspondentes a FSPs derivados de erros de mRNA. O desempenho do modelo sugere que esta abordagem pode fornecer um único teste para prever a resposta ao tratamento à ICI e identificar pacientes com alto risco de toxicidade ao tratamento.