 Danos ao DNA podem ser reconhecidos e reparados por vias de reparo de DNA em várias etapas, cada uma dedicada a tipos específicos de lesões e envolvendo muitos tipos de proteínas de reparo. O oncologista André Murad (foto) explica os sistemas de reparo do DNA, em mais um tópico da coluna ‘Drops de Genômica’. Confira.
Danos ao DNA podem ser reconhecidos e reparados por vias de reparo de DNA em várias etapas, cada uma dedicada a tipos específicos de lesões e envolvendo muitos tipos de proteínas de reparo. O oncologista André Murad (foto) explica os sistemas de reparo do DNA, em mais um tópico da coluna ‘Drops de Genômica’. Confira.
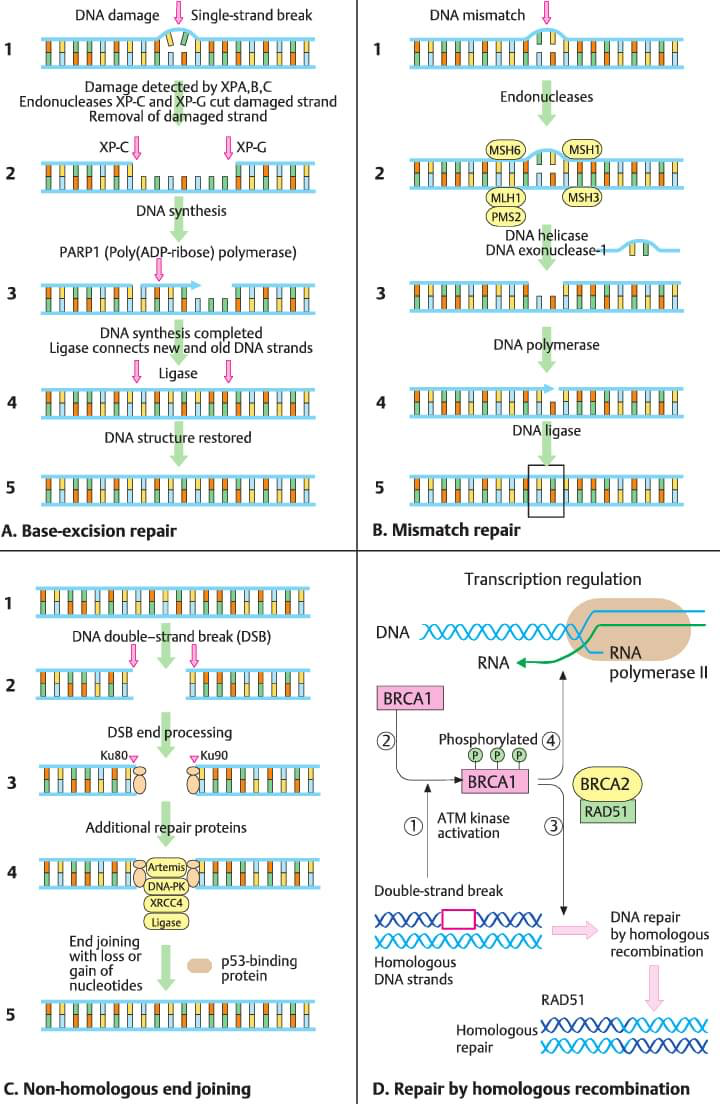
Os quatro tipos principais de reparo de DNA são: reparo por excisão de base, reparo de incompatibilidade, união de extremidades não homólogas e reparo de quebras de fita dupla.
A. Reparo de excisão de base
Este sistema remove 27 a 29 nucleotídeos da fita danificada. Ele reconhece uma fita de DNA distorcida, por exemplo, por dímeros de timina-timina provocados por luz ultravioleta. Três proteínas, XPA, XPB e XPC em células humanas detectam o local danificado e formam um complexo de proteínas de reparo. As endonudeases XP-C e XP-G clivam a fita danificada em dois locais. A síntese de reparo de DNA pela poli (ADP-ribose) polimerase 1 restaura o trecho ausente e uma ligase de DNA fecha a lacuna.
B. Reparo de incompatibilidade
Reparo de incompatibilidade é um sistema de ressíntese de excisão iniciado em uma quebra de DNA de 100 a 1.000 nucleotídeos de distância da incompatibilidade. Ele corrige erros de replicação. As proteínas de reparo de incompatibilidade mais importantes em humanos são MSH2, MSH3, MSH6; MLH1 e PMS2 (segregação pós-meiótica 2). O MSH2 e o MLH1 ligam-se a pares de bases incompatíveis, enquanto outros clivam o DNA e removem a fita com bases errôneas. A DNA polimerase substitui a fita danificada.
C. União de extremidades não homólogas
O reparo da fita dupla une as duas extremidades do DNA após uma quebra da fita dupla. As extremidades ligam-se a proteínas diméricas específicas de 80 e 90 kDa (Ku80 e Ku90). Proteínas de reparo adicionais formam um complexo de reparo, com uma proteína quinase dependente de DNA (DNA-PK), XRCC4 e uma ligação de proteína p53. Neste tipo de reparo, bases podem ser perdidas ou adicionadas.
D. Reparo de fita dupla por recombinação homóloga
A recombinação homóloga repara quebras de fita dupla usando várias proteínas de reparo: BRCA1, BRCA2, RAD5l, ATM, PALB2 e outras, como as proteínas de anemia de Fanconi. O ATM é ativado em resposta a danos no DNA. Isso ativa BRCA1 por fosforilação em locais específicos. BRCA1 fosforilado induz recombinação homóloga em cooperação com BRCA2 e RAD51, o homólogo de mamífero da proteína de reparo Escherlchla coll RecA. O BRCA1 fosforilado também está envolvido na transcrição e no reparo do DNA acoplado à transcrição. A cromátide irmã idêntica é usada para alinhar as pontas quebradas e inserir as informações que faltam.
Conceito de Letalidade Sintética (LS): surge quando uma combinação de deficiências na expressão de dois ou mais genes leva à apoptose celular, ao passo que a deficiência em apenas um desses genes não. As deficiências podem surgir por meio de mutações, alterações epigenéticas como metilação da região promotora dos genes ou por inibidores das enzimas PARP.
As enzimas PARP - poli (ADP-ribose) polimerase - estão envolvidas em uma via para reparar o DNA danificado dentro das células. Os genes BRCA (BRCA1 e BRCA2) e os demais que compõem o sistema de recombinação homóloga também estão normalmente envolvidos, mas em uma via diferente distinta, sendo que mutações nesses genes podem bloquear essa via. Ao bloquear a via PARP, esses medicamentos tornam difícil às células tumorais com um gene BRCA mutado reparar o DNA danificado, evento este que conduz essas células à apoptose, o que é denominada "letalidade sintética".
*André Murad é diretor científico do Grupo Brasileiro de Oncologia de Precisão (GBOP), diretor clínico da Personal - Oncologia de Precisão e Personalizada, professor adjunto coordenador da Disciplina de Oncologia da Faculdade de Medicina da UFMG, e oncologista e oncogeneticista da CETTRO Oncologia (DF)




