O oncologista André Murad (foto) aborda o versátil sistema CRISPR/Cas9 e como ele pode ser usado para modificar genomas com precisão e modular amplamente a expressão gênica. Confira, em mais uma coluna ‘Drops de Genômica’.
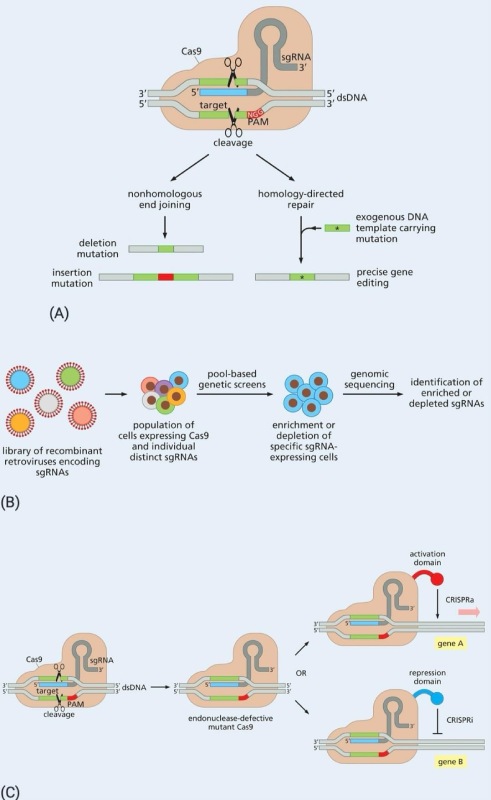
O versátil sistema CRISPR/Cas9 pode ser usado para modificar genomas com precisão e modular amplamente a expressão gênica. A endonuclease de DNA Cas9 derivada de S.pyogenes pode ser localizada em uma sequência de DNA específica por meio da ligação a um RNA de guia único (sgRNA) que também forma pares de bases por meio de uma sequência complementar a uma sequência alvo específica no genoma.
A sequência alvo deve ser adjacente a uma sequência de motivo adjacente protoespaçador (PAM), uma sequência de três nucleotídeos composta de qualquer nucleotídeo (N seguido por dois resíduos G (NGG). A clivagem mediada por Cas9 de ambas as fitas da sequência alvo resulta em uma quebra de DNA de fita dupla. O reparo da quebra pela via de união de extremidades não homólogas propensa a erros pode produzir mutações de exclusão e inserção, que normalmente causam perda da função do gene. Com a adição de um molde de DNA exógeno carregando uma mutação, o reparo direcionado por homologia pode transferir a mutação para o genoma.
Para realizar triagens genéticas baseadas em CRISPR, bibliotecas de retrovírus recombinantes são construídas, de modo que cada vírus carregue um sgRNA diferente visando a um gene específico. Esses vírus são usados para infectar células que expressam Cas9. A mutação subsequente mediada por CRISPR/Cas9 leva à geração de uma população de células com mutações direcionadas de perda de função nos genes correspondentes. Submeter essas células a condições seletivas causa enriquecimento ou depleção de certas células mutantes. Para triagens baseadas em enriquecimento, a identidade de mutações selecionadas pode ser determinada após sequenciamento de DNA das porções de sgRNA dos provírus retrovirais presentes nas células mutantes restantes. Para triagens baseadas em depleção, compara-se a representação de sequências de sgRNA na população inicial de células àquela presente após o crescimento nas condições seletivas; sequências de sgRNA que são relativamente sub-representadas na população final são indicativas de genes nos quais a mutação levou a uma desvantagem de aptidão.
Cas9 pode também ser transformado em uma endonuclease defeituosa por meio de mutações específicas. Essas proteínas mutantes Cas9 defeituosas em endonuclease ainda podem se ligar a sgRNAs e ser direcionadas a locais específicos no genoma. Ao fundir domínios de ativação ou repressão transcricional nas proteínas Cas9 com defeito na endonuclease e direcioná-las para as regiões promotoras de genes de interesse por meio da expressão dos sgRNAs apropriados, é possível afetar a ativação gênica mediada por CRISPR (CRISPRa) ou inibição (CRISPRi).
*André Murad é diretor científico do Grupo Brasileiro de Oncologia de Precisão (GBOP), diretor clínico da Personal - Oncologia de Precisão e Personalizada, professor adjunto coordenador da Disciplina de Oncologia da Faculdade de Medicina da UFMG, e oncologista e oncogeneticista da Clínica OncoLavras





